生物数学团队在组学数据建模和关键信息挖掘方面取得系列新进展
数学与统计学院生物数学团队在组学数据建模和统计分析方面取得系列进展。在国际顶级期刊《IEEE Journal of Biomedical and Health Informatics》上发表题为《TSPLASSO: A two-stage prior LASSO algorithm for gene selection using omics data》(DOI: 10.1109/JBHI.2023.3326485)的研究论文。该期刊中科院分区中为1区Top期刊,数学与统计学院2021级硕士研究生杨思佳是论文的第一作者,该工作在王沛教授指导下完成,博士生陈顺杰,陈爱敏副教授,田天海教授参与这项工作,河南大学是该论文的第一完成单位。
组学数据的特征选择已被广泛应用于识别癌症驱动基因。虽然研究者们已提出了一系列的组学数据挖掘方法来寻找癌症驱动基因,但已有方法较少考虑将已知的癌症驱动基因作为先验知识。该研究提出了一种融合先验信息的两阶段基因选择(TSPLASSO)方法。第一阶段基于“guilt-by-association”原则,将先验基因逐个作为响应,构建LASSO惩罚的线性回归模型,筛选出与已知癌症基因相关的候选基因用于后续分析。第二阶段将上述筛选出来的基因作为协变量,建立LASSO惩罚的逻辑回归模型,同时实现关键基因的筛选和样本的分类。在转录组、单细胞组等各类癌症组学数据中的仿真以及与多种已有方法的对比表明,TSPLASSO可以有效的选择癌症信息基因,TSPLASSO在一些转录组和单细胞组数据中将变量选择的精确度提高5%-400%;并且TSPLASSO对数据噪声和先验基因数量的变化具有鲁棒。
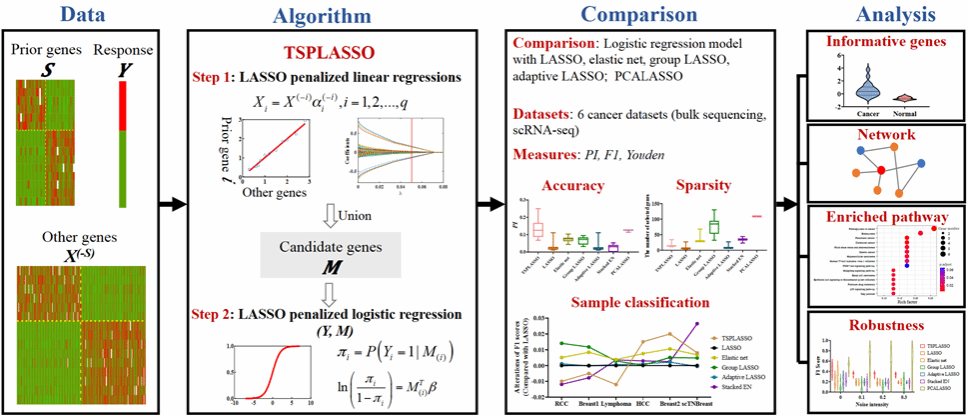
上述工作为融合先验信息的两阶段基因选择方法的一些早期尝试,为研究者们从组学数据中挖掘关键信息提供了有效的理论方法,具有潜在的生物医学应用前景和价值。
在学校和学院的大力支持下,生物数学团队近年来围绕高维生物医学组学数据分析、系统生物学、传染病建模与分析等领域的一些热点问题,在IEEE Trans.系列,Brief. Bioinformat., Inform Sci.,Nonlinear Dynamics, iScience, Physica A, BMC Plant Biol.等期刊上取得了系列成果,出版Springer专著1部,获河南省科技进步奖2项等。本研究工作得到了国家自然科学基金、河南省自然科学基金、河南省高校科技创新人才、河南大学青年科研创新团队等项目的资助。
IEEE Journal of Biomedical and Health Informatics论文网址:https://ieeexplore.ieee.org/document/10290898
